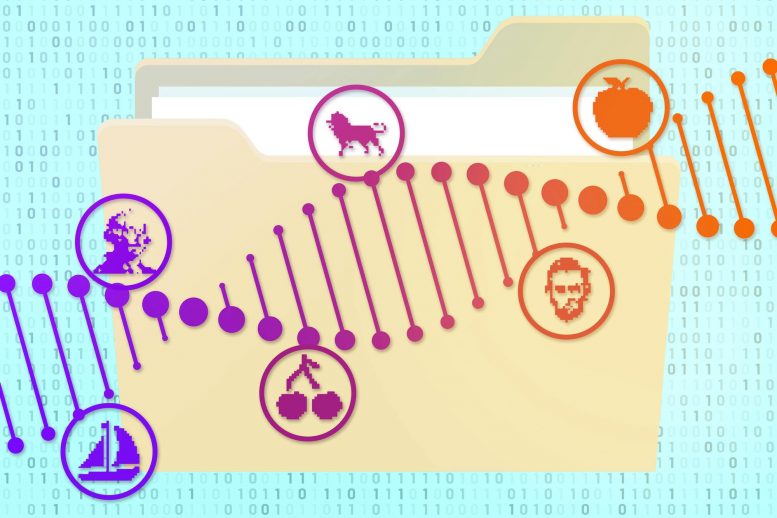
Les ingénieurs en biologie du MIT ont démontré un moyen de récupérer facilement les fichiers de données stockés sous forme d'ADN. Cela pourrait être une étape vers l'utilisation des archives ADN pour stocker d'énormes quantités de photos, d'images et d'autres contenus numériques. Crédit : Image : MIT News. Petites icônes avec l'aimable autorisation des chercheurs
Une technique d'étiquetage et de récupération des fichiers de données ADN à partir d'un grand pool pourrait aider à rendre possible le stockage des données ADN.
À l'heure actuelle, sur Terre, il existe environ 10 2.5 milliards de gigaoctets de données numériques, et chaque jour, les humains produisent des e-mails, des photos, des tweets et d'autres fichiers numériques qui totalisent 1 millions de gigaoctets de données supplémentaires. Une grande partie de ces données sont stockées dans d'énormes installations appelées centres de données exaoctets (un exaoctet équivaut à 1 milliard de gigaoctets), qui peuvent avoir la taille de plusieurs terrains de football et coûter environ XNUMX milliard de dollars à construire et à entretenir.
De nombreux scientifiques pensent qu'une solution alternative réside dans la molécule qui contient notre information génétique : l'ADN, qui a évolué pour stocker des quantités massives d'informations à très haute densité. Une tasse de café pleine d'ADN pourrait théoriquement stocker toutes les données du monde, explique Mark Bathe, professeur de génie biologique au MIT.
« Nous avons besoin de nouvelles solutions pour stocker ces énormes quantités de données que le monde accumule, en particulier les données d'archives », déclare Bathe, qui est également membre associé du Broad Institute du MIT et de Harvard. « L'ADN est mille fois plus dense que même la mémoire flash, et une autre propriété intéressante est qu'une fois que vous avez fabriqué le polymère d'ADN, il ne consomme aucune énergie. Vous pouvez écrire l'ADN et le stocker pour toujours.

Une photo des « fichiers » ADN. Chaque sphère de silice contient des séquences d'ADN qui codent pour une image particulière, et l'extérieur de la sphère est recouvert de codes-barres nucléotidiques qui décrivent le contenu de l'image. Crédit : Avec l'aimable autorisation des chercheurs
Les scientifiques ont déjà démontré qu'ils peuvent coder des images et des pages de texte sous forme d'ADN. Cependant, un moyen facile de sélectionner le fichier souhaité à partir d'un mélange de nombreux morceaux d'ADN sera également nécessaire. Bathe et ses collègues ont maintenant démontré une façon de le faire, en encapsulant chaque fichier de données dans une particule de silice de 6 micromètres, qui est étiquetée avec de courtes séquences d'ADN qui révèlent le contenu.
En utilisant cette approche, les chercheurs ont démontré qu'ils pouvaient extraire avec précision des images individuelles stockées sous forme de séquences d'ADN à partir d'un ensemble de 20 images. Compte tenu du nombre d'étiquettes possibles qui pourraient être utilisées, cette approche pourrait s'étendre jusqu'à 1020 fichiers.
Bathe est l'auteur principal de l'étude, qui paraît aujourd'hui dans Nature Materials. Les auteurs principaux de l'article sont le postdoctorant senior du MIT James Banal, l'ancien associé de recherche du MIT Tyson Shepherd et l'étudiant diplômé du MIT Joseph Berleant.
Stockage stable
Les systèmes de stockage numérique encodent le texte, les photos ou tout autre type d'informations sous la forme d'une série de 0 et de 1. Cette même information peut être codée dans l'ADN en utilisant les quatre nucléotides qui composent le code génétique : A, T, G et C. Par exemple, G et C pourraient être utilisés pour représenter 0 tandis que A et T représentent 1.
L'ADN a plusieurs autres caractéristiques qui le rendent souhaitable comme support de stockage : il est extrêmement stable et il est assez facile (mais coûteux) à synthétiser et à séquencer. De plus, en raison de sa densité élevée – chaque nucléotide, équivalent à jusqu'à deux bits, mesure environ 1 nanomètre cube – un exaoctet de données stockées sous forme d'ADN pourrait tenir dans la paume de votre main.
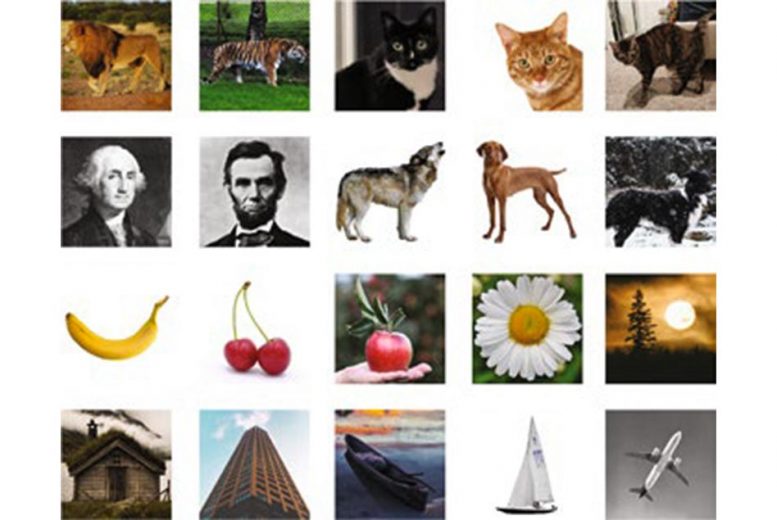
Les chercheurs ont stocké des images comme celles-ci, illustrées, dans l'ADN. Crédit : Avec l'aimable autorisation des chercheurs
Un obstacle à ce type de stockage de données est le coût de la synthèse d'aussi grandes quantités d'ADN. Actuellement, il en coûterait 1 1 milliards de dollars pour écrire un pétaoctet de données (XNUMX million de gigaoctets). Pour devenir compétitif avec la bande magnétique, qui est souvent utilisée pour stocker des données d'archives, Bathe estime que le coût de la synthèse d'ADN devrait baisser d'environ six ordres de grandeur. Bathe dit qu'il prévoit que cela se produira dans une décennie ou deux, de la même manière que le coût de stockage des informations sur les lecteurs flash a considérablement diminué au cours des deux dernières décennies.
Mis à part le coût, l'autre goulot d'étranglement majeur dans l'utilisation de l'ADN pour stocker des données est la difficulté de choisir le fichier que vous voulez parmi tous les autres.
« En supposant que les technologies d'écriture de l'ADN atteignent un point où il est rentable d'écrire un exaoctet ou un zettaoctet de données dans l'ADN, alors quoi ? Vous allez avoir une pile d'ADN, c'est-à-dire des milliards de fichiers, d'images ou de films et d'autres choses, et vous devez trouver la photo ou le film que vous recherchez », explique Bathe. "C'est comme essayer de trouver une aiguille dans une botte de foin."
Actuellement, les fichiers ADN sont classiquement récupérés par PCR (amplification en chaîne par polymérase). Chaque fichier de données ADN comprend une séquence qui se lie à une amorce PCR particulière. Pour extraire un fichier spécifique, cette amorce est ajoutée à l'échantillon pour trouver et amplifier la séquence souhaitée. Cependant, un inconvénient de cette approche est qu'il peut y avoir une interférence entre l'amorce et les séquences d'ADN hors cible, entraînant l'extraction de fichiers indésirables. De plus, le processus de récupération par PCR nécessite des enzymes et finit par consommer la majeure partie de l'ADN qui se trouvait dans le pool.
"Vous brûlez en quelque sorte la botte de foin pour trouver l'aiguille, car tout l'autre ADN n'est pas amplifié et vous le jetez essentiellement", explique Bathe.
Récupération de fichiers
Comme approche alternative, l'équipe du MIT a développé une nouvelle technique de récupération qui consiste à encapsuler chaque fichier d'ADN dans une petite particule de silice. Chaque capsule est étiquetée avec des « codes-barres » ADN simple brin qui correspondent au contenu du dossier. Pour démontrer cette approche de manière rentable, les chercheurs ont codé 20 images différentes en morceaux d'ADN d'environ 3,000 100 nucléotides de long, ce qui équivaut à environ XNUMX octets. (Ils ont également montré que les capsules pouvaient contenir des fichiers ADN allant jusqu'à un gigaoctet.)
Chaque fichier était étiqueté avec des codes-barres correspondant à des étiquettes telles que « chat » ou « avion ». Lorsque les chercheurs veulent extraire une image spécifique, ils retirent un échantillon de l'ADN et ajoutent des amorces qui correspondent aux étiquettes qu'ils recherchent - par exemple, "chat", "orange" et "sauvage" pour une image d'un tigre, ou « chat », « orange » et « domestique » pour un chat domestique.
Les amorces sont étiquetées avec des particules fluorescentes ou magnétiques, ce qui facilite l'extraction et l'identification de toutes les correspondances de l'échantillon. Cela permet de supprimer le fichier souhaité tout en laissant le reste de l'ADN intact pour être remis en stockage. Leur processus de récupération permet des déclarations logiques booléennes telles que « président ET 18th siècle" pour générer George Washington comme résultat, similaire à ce qui est récupéré avec une image Google recherche.
« Dans l'état actuel de notre preuve de concept, nous sommes au taux de recherche de 1 kilo-octet par seconde. Le taux de recherche de notre système de fichiers est déterminé par la taille des données par capsule, qui est actuellement limitée par le coût prohibitif pour écrire même 100 mégaoctets de données sur l'ADN, et le nombre de trieurs que nous pouvons utiliser en parallèle. Si la synthèse d'ADN devenait suffisamment bon marché, nous serions en mesure de maximiser la taille des données que nous pouvons stocker par fichier avec notre approche », explique Banal.
Pour leurs codes-barres, les chercheurs ont utilisé des séquences d'ADN simple brin d'une bibliothèque de 100,000 25 séquences, chacune d'environ 10 nucléotides, développée par Stephen Elledge, professeur de génétique et de médecine à la Harvard Medical School. Si vous mettez deux de ces étiquettes sur chaque fichier, vous pouvez étiqueter de manière unique XNUMX10 (10 milliards) de fichiers différents, et avec quatre étiquettes sur chacun, vous pouvez étiqueter de manière unique 1020 fichiers.
George Church, professeur de génétique à la Harvard Medical School, décrit la technique comme « un pas de géant pour la gestion des connaissances et la technologie de recherche ».
« Les progrès rapides de l'écriture, de la copie, de la lecture et du stockage de données d'archives à faible consommation d'énergie sous forme d'ADN ont laissé des opportunités mal explorées pour une récupération précise de fichiers de données à partir d'énormes (1021 octets, à l'échelle zetta) », explique Church, qui n'a pas participé à l'étude. "La nouvelle étude aborde ce problème de manière spectaculaire en utilisant une couche externe d'ADN complètement indépendante et en tirant parti de différentes propriétés de l'ADN (hybridation plutôt que séquençage), et de plus, en utilisant des instruments et des chimies existants."
Bathe envisage que ce type d'encapsulation d'ADN pourrait être utile pour stocker des données « froides », c'est-à-dire des données conservées dans des archives et peu consultées. Son laboratoire est en train de lancer une startup, Cache DNA, qui développe actuellement une technologie pour le stockage à long terme de l'ADN, à la fois pour le stockage des données ADN à long terme et des échantillons d'ADN cliniques et autres préexistants à court terme.
« Bien qu'il puisse s'écouler un certain temps avant que l'ADN ne soit viable en tant que support de stockage de données, il existe déjà aujourd'hui un besoin pressant de solutions de stockage massives et à faible coût pour les échantillons d'ADN et d'ARN préexistants provenant des tests Covid-19, du séquençage génomique humain et domaines de la génomique », dit Bathe.
Référence : « Random access DNA memory using Boolean search in an archive file storage system » par James L. Banal, Tyson R. Shepherd, Joseph Berleant, Hellen Huang, Miguel Reyes, Cheri M. Ackerman, Paul C. Blainey et Mark Bathe, 10 juin 2021, Nature Materials.
DOI: 10.1038/s41563-021-01021-3
La recherche a été financée par l'Office of Naval Research, la National Science Foundation et le US Army Research Office.









