Gli ingegneri biologici del MIT hanno dimostrato un modo per recuperare facilmente i file di dati archiviati come DNA. Questo potrebbe essere un passo verso l'utilizzo degli archivi del DNA per archiviare enormi quantità di foto, immagini e altri contenuti digitali. Credito: Immagine: MIT News. Piccole icone per gentile concessione dei ricercatori
Una tecnica per etichettare e recuperare i file di dati del DNA da un grande pool potrebbe contribuire a rendere fattibile l'archiviazione dei dati del DNA.
Sulla Terra in questo momento ci sono circa 10 trilioni di gigabyte di dati digitali e ogni giorno gli esseri umani producono e-mail, foto, tweet e altri file digitali che aggiungono fino a 2.5 milioni di gigabyte di dati. Gran parte di questi dati è archiviata in enormi strutture note come data center exabyte (un exabyte equivale a 1 miliardo di gigabyte), che possono avere le dimensioni di diversi campi da calcio e la costruzione e la manutenzione costano circa 1 miliardo di dollari.
Molti scienziati ritengono che una soluzione alternativa risieda nella molecola che contiene le nostre informazioni genetiche: il DNA, che si è evoluto per immagazzinare enormi quantità di informazioni ad altissima densità. Una tazza di caffè piena di DNA potrebbe teoricamente memorizzare tutti i dati del mondo, afferma Mark Bathe, professore di ingegneria biologica del MIT.
"Abbiamo bisogno di nuove soluzioni per archiviare queste enormi quantità di dati che il mondo sta accumulando, in particolare i dati d'archivio", afferma Bathe, che è anche membro associato del Broad Institute del MIT e di Harvard. “Il DNA è mille volte più denso persino della memoria flash, e un'altra proprietà interessante è che una volta prodotto il polimero del DNA, non consuma energia. Puoi scrivere il DNA e poi conservarlo per sempre”.

Una foto dei "file" del DNA Ogni sfera di silice contiene sequenze di DNA che codificano un'immagine particolare e l'esterno della sfera è rivestito con codici a barre nucleotidici che descrivono il contenuto dell'immagine. Credito: per gentile concessione dei ricercatori
Gli scienziati hanno già dimostrato di poter codificare immagini e pagine di testo come DNA. Tuttavia, sarà necessario anche un modo semplice per selezionare il file desiderato da una miscela di molti pezzi di DNA. Bathe e i suoi colleghi hanno ora dimostrato un modo per farlo, incapsulando ogni file di dati in una particella di silice di 6 micrometri, etichettata con brevi sequenze di DNA che ne rivelano il contenuto.
Utilizzando questo approccio, i ricercatori hanno dimostrato di poter estrarre con precisione singole immagini memorizzate come sequenze di DNA da un set di 20 immagini. Dato il numero di possibili etichette che potrebbero essere utilizzate, questo approccio potrebbe scalare fino a 1020 File.
Bathe è l'autore senior dello studio, che appare oggi in Nature Materials. Gli autori principali del documento sono il postdoc senior del MIT James Banal, l'ex ricercatore associato del MIT Tyson Shepherd e lo studente laureato del MIT Joseph Berleant.
Stoccaggio stabile
I sistemi di archiviazione digitale codificano testo, foto o qualsiasi altro tipo di informazione come una serie di 0 e 1. Questa stessa informazione può essere codificata nel DNA usando i quattro nucleotidi che compongono il codice genetico: A, T, G e C. Ad esempio, G e C potrebbero essere usati per rappresentare 0 mentre A e T rappresentano 1.
Il DNA ha molte altre caratteristiche che lo rendono desiderabile come supporto di memorizzazione: è estremamente stabile ed è abbastanza facile (ma costoso) da sintetizzare e sequenziare. Inoltre, a causa della sua alta densità - ogni nucleotide, equivalente a un massimo di due bit, è di circa 1 nanometro cubo - un exabyte di dati archiviati come DNA potrebbe stare nel palmo della tua mano.
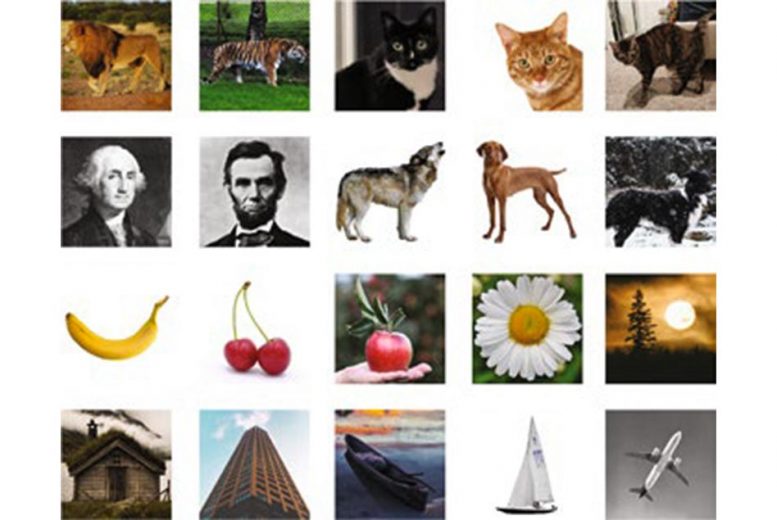
I ricercatori hanno memorizzato immagini come queste, raffigurate, nel DNA. Credito: per gentile concessione dei ricercatori
Un ostacolo a questo tipo di memorizzazione dei dati è il costo della sintesi di così grandi quantità di DNA. Attualmente costerebbe 1 trilione di dollari scrivere un petabyte di dati (1 milione di gigabyte). Per diventare competitivo con il nastro magnetico, che viene spesso utilizzato per archiviare dati d'archivio, Bathe stima che il costo della sintesi del DNA dovrebbe diminuire di circa sei ordini di grandezza. Bathe afferma di prevedere che ciò accadrà entro un decennio o due, in modo simile a come il costo di archiviazione delle informazioni su unità flash è diminuito drasticamente negli ultimi due decenni.
A parte il costo, l'altro grosso collo di bottiglia nell'uso del DNA per archiviare i dati è la difficoltà nel selezionare il file desiderato da tutti gli altri.
“Supponendo che le tecnologie per scrivere il DNA arrivino a un punto in cui è conveniente scrivere un exabyte o uno zettabyte di dati nel DNA, allora cosa? Avrai un mucchio di DNA, che è un fantastiliardo di file, immagini o film e altre cose, e devi trovare l'immagine o il film che stai cercando”, dice Bathe. "È come cercare di trovare un ago in un pagliaio."
Attualmente, i file di DNA vengono recuperati convenzionalmente mediante PCR (reazione a catena della polimerasi). Ogni file di dati del DNA include una sequenza che si lega a un particolare primer PCR. Per estrarre un file specifico, quel primer viene aggiunto al campione per trovare e amplificare la sequenza desiderata. Tuttavia, uno svantaggio di questo approccio è che può esserci diafonia tra il primer e le sequenze di DNA fuori bersaglio, portando all'estrazione di file indesiderati. Inoltre, il processo di recupero della PCR richiede enzimi e finisce per consumare la maggior parte del DNA che era nel pool.
"Stai bruciando il pagliaio per trovare l'ago, perché tutto il resto del DNA non viene amplificato e praticamente lo stai buttando via", dice Bathe.
Recupero file
Come approccio alternativo, il team del MIT ha sviluppato una nuova tecnica di recupero che prevede l'incapsulamento di ogni file di DNA in una piccola particella di silice. Ogni capsula è etichettata con "codici a barre" di DNA a filamento singolo che corrispondono al contenuto del file. Per dimostrare questo approccio in modo conveniente, i ricercatori hanno codificato 20 diverse immagini in pezzi di DNA lunghi circa 3,000 nucleotidi, che equivalgono a circa 100 byte. (Hanno anche dimostrato che le capsule possono contenere file di DNA di dimensioni fino a un gigabyte.)
Ogni file è stato etichettato con codici a barre corrispondenti a etichette come "gatto" o "aereo". Quando i ricercatori vogliono estrarre un'immagine specifica, rimuovono un campione del DNA e aggiungono primer che corrispondono alle etichette che stanno cercando, ad esempio "gatto", "arancione" e "selvaggio" per un'immagine di una tigre, o "gatto", "arancione" e "domestico" per un gatto domestico.
I primer sono etichettati con particelle fluorescenti o magnetiche, facilitando l'estrazione e l'identificazione di eventuali corrispondenze dal campione. Ciò consente di rimuovere il file desiderato lasciando intatto il resto del DNA da riporre in deposito. Il loro processo di recupero consente affermazioni logiche booleane come "presidente AND 18th secolo” per generare come risultato George Washington, simile a quello recuperato con un’immagine di Google Ricerca.
“Allo stato attuale del nostro proof-of-concept, siamo alla velocità di ricerca di 1 kilobyte al secondo. La velocità di ricerca del nostro file system è determinata dalla dimensione dei dati per capsula, che è attualmente limitata dal costo proibitivo per scrivere anche 100 megabyte di dati sul DNA, e dal numero di selezionatori che possiamo usare in parallelo. Se la sintesi del DNA diventasse abbastanza economica, con il nostro approccio saremmo in grado di massimizzare la dimensione dei dati che possiamo archiviare per file", afferma Banal.
Per i loro codici a barre, i ricercatori hanno utilizzato sequenze di DNA a filamento singolo da una libreria di 100,000 sequenze, ciascuna lunga circa 25 nucleotidi, sviluppata da Stephen Elledge, professore di genetica e medicina alla Harvard Medical School. Se metti due di queste etichette su ciascun file, puoi etichettare in modo univoco 1010 (10 miliardi) di file diversi e con quattro etichette su ciascuno, puoi etichettare in modo univoco 1020 File.
George Church, professore di genetica alla Harvard Medical School, descrive la tecnica come "un passo da gigante per la gestione della conoscenza e la tecnologia di ricerca".
“Il rapido progresso nella scrittura, copia, lettura e archiviazione dei dati di archiviazione a bassa energia in forma di DNA ha lasciato opportunità poco esplorate per il recupero preciso di file di dati da enormi (1021 byte, zetta-scale)”, dice Church, che non è stata coinvolta nello studio. “Il nuovo studio affronta in modo spettacolare questo utilizzando uno strato esterno di DNA completamente indipendente e sfruttando diverse proprietà del DNA (ibridazione piuttosto che sequenziamento) e, inoltre, utilizzando strumenti e prodotti chimici esistenti”.
Bathe prevede che questo tipo di incapsulamento del DNA potrebbe essere utile per memorizzare dati "freddi", cioè dati che vengono conservati in un archivio e non vi si accede molto spesso. Il suo laboratorio sta avviando una startup, Cache DNA, che ora sta sviluppando una tecnologia per l'archiviazione a lungo termine del DNA, sia per l'archiviazione dei dati del DNA a lungo termine, sia per campioni di DNA clinici e preesistenti a breve termine.
"Anche se potrebbe volerci un po' di tempo prima che il DNA sia praticabile come supporto di archiviazione dati, esiste già oggi un'urgente necessità di soluzioni di archiviazione massicce e a basso costo per campioni di DNA e RNA preesistenti provenienti da test Covid-19, sequenziamento genomico umano e altri aree della genomica", afferma Bathe.
Riferimento: "Random access DNA memory using Boolean search in an archival file storage system" di James L. Banal, Tyson R. Shepherd, Joseph Berleant, Hellen Huang, Miguel Reyes, Cheri M. Ackerman, Paul C. Blainey e Mark Bathe, 10 giugno 2021, Nature Materials.
DOI: 10.1038/s41563-021-01021-3
La ricerca è stata finanziata dall'Office of Naval Research, dalla National Science Foundation e dall'US Army Research Office.









